Enzim kinetiği enzimler tarafından katalizlenen kimyasal reaksiyonların bilimidir. Enzim kinetiğinde reaksiyon hızı ölçülür ve reaksiyon şartlarını değiştirmenin etkisi araştırılır. Bir enzimin kinetiğinin bu şekilde çalışılması enzimin katalitik mekanizmasını, metabolizmadaki rolünü, aktivitesinin nasıl kontrol edildiğini ve bir ilaç veya zehrin enzimi nasıl edebileceğini ortaya koyabilir.

Enzimler genelde diğer molekülleri (enzimin substratları) manipüle eden protein molekülleridir. Bu hedef moleküller enzimin aktif bölgesine bağlanır ve, enzim mekanizması denen bir seri adımlar sonucunda, ürünlere dönüşür. Bu mekanizmalar tek substratlı ve çok substratlı mekanizmalar olarak ayrılabilir. Tek bir substrata bağlanan enzimler ( gibi) üzerinde yapılan kinetik çalışmalar, enzimin substratına bağlanma (ilgisini) ve devir hızını ölçmeyi amaçlar.
Enzimler birden çok substrata bağlanınca, (sağda gösterilen) gibi, enzim kinetiği bu substratların bağlanma sırasını ve ürünlerin salınma sırasını gösterebilir. Bir tek substrata bağlanıp birden çok ürün salan enzimlere örnek, proteazlardır, bunlar bir protein substratı kesip iki polipeptit ürün oluştururlar. Başka enzimler iki substratı birleştirir, öreneğin DNA polimeraz'ın DNA'ya bir nükleotit eklemesi gibi. Bu mekanizmalar genelde karmaşık bir adımlar dizisi olsa da, tipik olarak bir hız belirleyici basamak, tüm reaksiyonun hızını belirler. Bu kimyasal bir reaksiyon veya, enzim veya substratlarında konformasyonel bir değişim olabilir, ürünlerin enzimden salınması sırasında görüldüğü gibi.
Enzimin yapısının bilinmesi kinetik verilerin yorumlanmasında faydalıdır. Örneğin, yapının bilinmesi sayesinde substrat ve ürünlerin kataliz sırasında nasıl bağlandıkları, reaksiyon sırasından hangi değişikliklerin meydana geldiği ve hatta mekanizmadaki belli amino asit kalıntılarınını rolleri tahmin edilebilir. Bazı enzimler mekanizma sırasında önemli derecede biçimlerini değiştirirler; böylesi durumlarda enzimin tek başına yapısının ve enzimatik reaksiyona girmeyen substrat analogları ona bağlı ikenki yapısının belirlenmesi yararlı olur.
Tüm biyolojik katalizörler protein değildir; ribozim ve ribozom gibi RNA-temelli katalizörler pek çok hücresel işlev için esastırlar, örneğin RNA uçbirleştirmesi (splicing) ve çeviri (translasyon) gibi. Ribozimler ve enzimler arasındaki temel fark, RNA katalizörlerin nükleotitlerden, enzimlerin ise amino asitlerden oluşmasıdır. Ribozimler daha sınırlı bir reaksiyonlar grubunu katalizler, ama reaksiyon mekanizmaları ve kinetikleri protein enzimleri ile aynı yöntemlerle analiz edilebilir ve sınıflandırılabilir.
Genel ilkeler

Bir enzim tarafından katalizlenen reaksiyon, katalizlenmeyen reaksiyon ile aynı denge özelliklerine sahiptir. Diğer katalizörler gibi, enzimler substrat ve ürünler arasındaki dengenin konumunu değiştirmezler. Ancak, katalizlenmeyen bir kimyasal reaksiyondan farklı olarak, enzimle katalizlenen reaksiyonlar doyum kinetiği gösterirler. Belli bir enzim konsantrasyonu ve nispeten düşük substrat konsantrasyonu için, reaksiyon hızı substrat konsantrasyonu ile doğrusal orantılı olarak artar; enzim moleküllerinin çoğu, reaksiyonu katalizlemek için müsaittir ve substrat konsantrasyonun artırılması, enzim ve substrat moleküllerinin birbirine rastlama hızının artması demektir. Ancak, nispeten yüksek substrat konsantrasyonlarıda, reasiyon hızı asimptotik şekilde teorik maksimuma yaklaşır; enzim aktif bölgelerinin hemen hepsi doludur ve reaksiyon hızını, enzimin kendine has (içsel) devir hızı belirler. Bu iki sınır durumun ortasındaki substrat konsantrasyonu KM olarak belirtilir. Bir diğer deyişle, en yüksek reaksiyon hızının (Vmax) yarısının elde edildiği substrat konsantrasyonu KM olarak tanımlanır.
Bir enzimin en önemli iki kinetik özelliği, enzimin belli bir substrat ile ne kadar çabuk doyuma ulaştığı ve ulaşabildiği en hızlı reaksiyon hızıdır. Bu özelliklerin bilinmesi bir enzimin hücre içinde ne yaptığını ve o şartlardaki değişiklere nasıl tepki verdiği hakkında fikir verir.
Enzim ölçümleri

Enzim ölçümleri enzim reakasiyonunun hızını ölçen laboratuvar yöntemleridir. Enzimler katalizledikleri reaksiyonlar tarafından tüketilmediği için, enzim ölçümlerinde reaksiyon hızını takip etmek için genelde ya substrat konsantrasyonu ya da ürün konsantrasyonu izlenir. Ölçüm için pek çok yöntem vardır. ölçümlerde substrat ve ürünlerdeki ışık değişim gözlemlenir; radyometrik ölçümlerde radyoaktivitenin bir ürüne dahil oluşu veya substrattan ayrılmasına bakarak zamana göre ürün oluşumunu ölçülür. Spektrofotometrik ölçümler en kullanışlıdır, çünkü reaksiyon hızının sürekli olarak ölçülmesi mümkündür. Radyometrik ölçümler numunelerin alınıp onların ölçümünü gerektirse de (yani kesintili ölçümlerdir) genelde daha duyarlıdırlar ve çok düşük enzim aktivite düzeyleri ölçülebilir. Benzer bir yaklaşım, kütle spektrometresi kullanarak, ürüne stabil bir izotopun enkorpore olmasının veya substrattan salınmasının izlenmesidir.
En duyarlı enzim ölçümleri bir mikroskop içinden odaklanmış bir lazer kullanarak reaksiyonunu katalizleyen tek bir enzim molekülünü gözlemler.Bu ölçümler enzimin reaksiyon mekanizması sırasında kofaktörlerin floresansındaki değişimi veya protein üzerinde belli konumlara bağlanmış değişimi ölçer, kataliz sırasındaki hareketleri izlemek için. Milyonlarca enzim molekülünün ortalama davranışını gözlemleyen geleneksel enzim kinetiğine karşın, bu tür çalışmalar tekil enzimlerin kinetiği ve dinamiği hakkında yeni bir bakış açısı sağlar.
Bir enzim ölçümü için bir ilerleme eğrisi örneği yukarıda gösterilmiştir. Enzim, reaksiyonun başlamasında sonra kısa bir süre için doğrusal şekilde ürün meydana getirir. Reaksiyon ilerledikçe ve substrat tüketildikçe, hızı gittikçe azalır (substrat doyum seviyesinde olmadıkça). İlk (ve en yüksek) hızı ölçmek için, enzim ölçümleri tipik olarak substratın sadece yüzde birkaçı tüketilene kadar yapılır. Bu ilk hız döneminin uzunluğu ölçüm şartlarına bağlıdır ve milisaniyeleden saatlere kadar değişebilir. Ancak, sıvıların çok hızlı karşmasını sağlayan araçlar, bir sanıyeden kısa sürelerde ilk hızların ölçülebilmesini olanak verir. Aşağıda anlatıldığı üzere, bu çok hızlı ölçümler, sabit durum öncesi (pre-steady state) kinetiğinin ölçülmesi için şarttır.
Çoğu enzim kinetik çalışmaları enzim reaksiyonlarının bu ilk, yaklaşık doğrusal, kısmı üzerine yoğunlaşır. Ancak, tüm reaksiyonu ölçüp bu verileri doğrusal olmayan bir uydurmak da mümkündür. Enzim reaksiyonlarının bu şekilde ölçülmesi, ilerleme eğrisi analizi (İng. progress-curve analysis) olarak adlandırılır. Eğer ilk hız, ile doğru ölçülemeyecek kadar yüksek ise, bu yaklaşım yararlı bir alternatiftir.
Tek substrat reaksiyonları
Tek substrat mekanizmalı enzimlere örnek olarak, izomerazlar ( veya gibi), gibi hücre içi liyazlar ve bir olan sayılabilir. Ancak, tek bir substratı olan bazı enzimler bu mekanizma kategorisi içine girmezler. Bunun bir örneği olan katalaz, substratı olan hidrojen peroksit ile reaksiyona girince yükseltgenir (okside olur) ve sonra ikinci bir substrat molekülü tarafından indirgenir. Substrat tek olsa da, modifiye olmuş bir enzim ara ürününün varlığı, katalaz mekanizmasının aslında bir pinpon (masa tenisi) mekanizması olduğunu gösterir. Bu mekanizma tipi aşağıda Çok substratlı reaksiyonlar bölümü altında açıklanmıştır.
Michaelis-Menten kinetiği


Enzimle katalizlenmiş reaksiyonlar doyumlu olduğu için, kataliz hızları artan substrata doğrusal bir tepki göstermez. Eğer ilk reaksiyon hızı çeşitli substrat konsantrasyonları ([S] ile gösterilir) için ölçülürse, reaksiyon hızı (v) [S] arttıkça artar, sağda gösterildiği gibi. Ancak, [S] yükseldikçe, enzim doygunlaşır (satüre olur) ve hız, enzimin en büyük hızı olan Vmax'a ulaşır.
Tek substratlı reaksiyon için Michaelis-Menten kinetik modeli sağda gösterilmiştir. Enzim E ve substratı S arasında önce bir iki moleküllü reaksiyon olup bir enzim-substrat reaksiyonu ES meydana gelir. Tek moleküllü reaksiyon 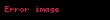
Eğer reaksiyon yolu bir veya birkaç araürün üzerinden gidiyorsa, kcat birkaç öğesel (elementer) hız sabitinin fonsiyonu olacaktır. Oysa en basit durum olan tek bir öğesel reaksiyonda (yani araürün olmaması hâlinde), öğesel tek moleküllü sabit k2'ye eşit olacaktır. Görünür tek-moleküllü hız sabiti kcatdevir hızı olarak da adlandırılır ve bu, saniyedeki katalizlenen en yüksek enzim reaksiyon sayısına karşılık gelir.
Michaelis–Menten denklemi (başlangıç) reaksiyon hızı v0'nin substrata bağlanma denge konumuna ve hız sabiti k2'ye nasıl bağlı olduğunu betimler.
(Michaelis–Menten denklemi)
sabitler:
Bu Michaelis-Menten denklemi çoğu tek substratlı enzim kinetiğinin temelinde yatar. Mekanizma hakkında genel bir varsayım, araürün veya ürün inhibisyonu olmadığı ve veya olmadığıdır. Bu denklemin altında yatan iki önemli varsayım daha vardır. Birincisi, sabit-durumumsuluk varsayımı (İng. quasi-steady-state assumption) (veya sahte sabit durum hipotezi) olarak adlandırılır, substrata bağlı enzim konsantrasyonundaki (ve dolayısıyla bağlı olmayan enzimin konsantrasyonundaki de) değişim hızının, ürün ve substratın konsantrasyonlarındaki değişmeden çok daha az olduğudur, dolayısıyla kompleksteki değişimin zamana bağlı değişiminin sıfıra eşitlenebileceğidir. 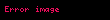
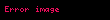
Michaelis sabiti KM'nin deneysel tanımı, enzim reaksiyon hızının Vmax'ın yarısı olduğu substrat konsantrasyonudur. Michaelis–Menten denkleminde [S] = Km substitusyonu yapılarak bu tanım doğrulanabilir, ayrıca grafik olarak da gösterilebilir. Eğer hız belirleyici enzimatik adım, substrat ayrışma hızına (
Eğer 
Yani, ürün oluşma hızı hem enzim konsantrasyonuna, hem de substrat konsantrasyonuna bağlıdır; psödo-ikinci derece hız sabiti 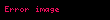
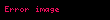
Michaelis-Menten denkleminin zaman bağımlı kinetik analizi için kullanımı
Michaelis-menten denklemi tarafından öngörülen reaksiyon hızlarından yararlanılarak zaman bağımlı substrat yokoluşu ve ürün oluşumu doğrudan modellenebilir. Bunu yapmak için birinci derece kimyasal kinetik denklemi içine Michaelis-Menten denklemi dahil edilir. Birinci derece kimayasal kinetiğin tarifinde Euler sayısının kullanımı ile ilgili problemin farkına varmak gerekir, yani e-k hesaplamalara sistematik bir hata sokan bir ayrık katsayıdır. Bu, tek bir katsayı kullanılarak yeniden yazılabilir, bu yeni katsayı her zaman aralığından sonraki kalan substrat miktarına karşılık gelir.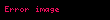
Michaelis–Menten denklemi için doğrusal grafikler

Yukarıda gösterilen, [S]'ye bağlı v grafiği doğrusal değildir; grafik, düşük [S] için başlangıçta doğrusal olsa dahi, yüksek [S]'de eğilerek doyuma ulaşır. Bu konuda yapılan ilk çalışmalarda, bu nonlineerlik KM ve Vmax'ın hatasız olarak kestirilmesini zor kılmaktaydı. Bu yüzden, bazı araştırmacılar Michaelis-Menten denkleminin lineerleştiren grafik yöntemler geliştirdiler: , ve gibi. Bu lineer gösterimlerin hepsi verinin görüntülenmesi için yararlıdır, ama kinetik parametrelerinin belirlenmesi için günümüzde bilgisayar yazılımları mevcuttur ve nonlineer regresyon yöntemleri kullanarak daha hatasız sonuçlar elde edilmesi mümkündür.
veya çifte evrik (İng. reciprocal) grafiği kinetik veriyi görüntülemek için yaygın kullanılır. Bunu elde etemk için Michaelis-Menten denkleminin iki tarafının yapılır. Sağda gösterildiği gibi, bu, Michaelis-Menten denkleminin doğrusal bir biçimidir ve y = mx + c denklemli bir doğru üretir, y ekseni kesişim noktası 1/Vmax'ye x eksen kesişimi -1/KM'ye karşılık gelir.
Tabii ki, negatif 1/[S] değerleri için deneysel değerler elde edilemez; 1/[S] = 0 alt sınırı (y kesişimi) sonsuz substrat konsantrasyonuna karşılık gelir, orada 1/v=1/Vmax'dır, sağda gösterildiği gibi. Dolayısıyla x-kesişimi pozitif konsantrasyonlarda elde edilen deneysel verilerin bir ekstrapolasyonudur. Daha genel olarak, Lineweaver–Burk grafiği düşük konsantrasyonlarda elde edilen ölçümlerin önemini çarpıtır ve dolayısıyla Vmax ve KM'nin hatalı kestirilmesine yol açabilir. Daha hatasız bir lineer grafikleme yöntemi . Bu durumda v, v/[S]'ye bağlı olarak grafiklenir. Yaygın kullanılan yöntemlerden üçüncüsü olan , [S]/v, [S]'ye bağlı olarak grafiklenir. Genelde, data normalizasyonu deneysel iş miktarını azaltmaya yarar, sonucun güvenilirliğini artırabilir ve hem grafik hem de sayısal analiz için uygundur.
Kinetik sabitlerin pratik anlamı
Enizm kinetiğinin araştırılması iki temel nedenden dolayı önemlidir. Birincisi, enzimlerin nasıl çalıştığının anlaşılmasını sağlar, ikincisi, enzimlerin organizmalarda nasıl davrandığını öngörmeye yardımcı olur. Yukarıda tanımlanan kinetik katsayılar KM ve Vmax, metabolima kontrolünde enzimlerin nasıl çalıştığının anlaşılmasında önemli rol oynar.
Bu öngörülerin yapılması sıradan değildir, basit sistemler için dahi. Örneğin, , mitokondride bulunan tarafından meydana gelir. Oksaloasetat sonra , veya tarafından tüketilebilir ve, sırasıyla, sitrik asit döngüsü, veya aspartik asit biyosentezini besleyebilir. Bu yolaklardan her birine ne kadar oksaloasetat gittiğini öngörmek için oksaolasetat konsantrasyonunu bilmek, ayrıca bu enzimlerin konsantrasyon ve kinetik özelliklerini bilmek gerekir. Metabolik yolakların davranışlarını öngörüsünün en karmaşık kısmı, tüm bir organizmaya ait matematiksel modellerin içine girmesi gereken kinetik ve gen ifadesi verilerinin sentezini yapılmasındadır. Buna alternatif olarak, kinetik metabolizma probleminin faydalı bir sadeleştirmesi, enzim kinetiklerini dikkate almayıp sadece reaksiyon ağının stokyometresi hakkındaki bilgileri dayanmaktır, bu yöntem olarak adlandırılır.
Ararürünlü Micahelis-Menten kinetiği
Basit olan şu durum ile başlayalım:
burada bir enzim ve araürün mevcuttur, araürün ikinci adımda ürüne dönüşür. Bu durumda çok benzer bir denklem ortaya çıkar.
Sınırlayıcı durum olan 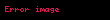
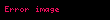

Çok-substratlı reaksiyonlar
Çok substratlı reaksiyonlarda subsratların nasıl bağlandığını ve hangi sırayla bağlandığını belirten karmaşık hız denklemleri vardır. Eğer A substratının konsantrasyonu sabit tutulur ve B substratınınki değiştirilirse, bu reaksiyonların analizi çok basitleşir. Bu şartlar altında, enzim tek substratlı bir enzim gibi davranış gösterir, v'yi [S]'ye bağlı olarak grafiklendiğinde, B substatı için görünür KM ve Vmax sabitleri elde edilir. Eğer bu ölçümler A'nın farklı sabit konsantrasyonları için yapılırsa, bu veriler kullanılarak reaksiyonun mekanizması anlaşılabilir. İki substrat A ve B'yi alıp bunları iki ürün P ve Q'ya dönüştüren bir enzim için iki tip mekanizma vardır, üçlü kompleks veya pinpon.
Üçlü kompleks mekanizmaları

Bu enzimlerde, her iki substrat enzime aynı anda bağlanıp bir EAB üçlü kompleksi meydana getirir. Bağlanma sırası rastgele olabilir (rastgele mekanizmada) veya substratların belli bir sıra sıra ile bağlanması gerekebilir (sıralı mekanizmada). Üçlü kompleks mekanizmalı bir enzim için bir v-[S] eğriler kümesi (sabit A, değişen B) bir Lineweaver-Burk grafiğinde çizilirse, doğrular bir noktada kesişir.
Üçlü kompleks mekanizmalı enzimler arasında , and sayılabilir. Dihidrofoloat redüktaz ve DNA polimeraz için üçlü-kompleks mekanizmalarını gösteren kısa animasyonlar şu bağlantıda izlenebilir.{{Ref_label|B|β|none}[γ]
Pinpon mekanizması

Sağda gösterildiği gibi, pinpon mekanizmalı enzimler iki durumda bulunabilir: asıl hâlleri olan E ve kimyasal olarak modifiye bir hâl olan E*. Enzimin bu modifiye olmuş hâli "" olarak adlandırılır. Bu mekanizmada, substrat A enzime bağlanır, aktif bölgeye bir kimyasal grup aktararak, enzimi E*'a dönüştürür ve sonra enzimden salınır. İlk substrat salındıktan sonra ancak substrat B bağlanabilir ve modifiye olmuş enzimle reaksiyona girip modifiye olmamış E biçimini yeniden oluşturur. Pinpon mekanizmalı bir enzim için bir v-[S] eğriler kümesi (sabit A, değişen B), Lineweaver-Burk grafiğinde çizilirse, doğrular bir noktada kesişir. Bu grafik, olarak adlandırılır.
Pinpon mekanizmalı enzimler arasında bazı oksidoredüktazlar (tiyoredoksin peroksidaz gibi), (asilnöraminat citidililtransferaz gibi) ve (tripsin ve gibi) sayılabilir. Serin proteazlar yaygın ve çeşitli üyelere sahib bir enzim ailesidir, aralarında sindirici enzimler (tripsin, kimotripsin ve elastaz), kan pıhtılaşma kaskadının bazı enzimleri ve diğerleri sayılabilir. Serin proteazlarda, E* araürünü bir asil-enzim türüdür, aktif bölge serin kalınıtısının bir protein substratının peptit bağına saldırısı sonucu meydana gelir. Kemotripsin mekanizmasını gösteren bir kısa animasyona bir bağlantı şurada verilmiştir[δ]
Michaelis–Menten dışı kinetikler

Bazı enzimlerin v - [S] grafiği şeklindedir, bu şekilli grafik, substratın aktif bölgeye bağlanmasında olduğunun belirtisidir. Bu demektir ki, bir substratın bağlanması sonraki substrat moleküllerinin bağlanmasına etki eder. Bu davranış, birbiriyle etkleşen birkaç aktif bölgenin olduğu mültimerik proteinlerde yaygındır. Kooperasyon mekanizması, hemoglobinde görülen bağlanma sürecine benzer, bir substratın aktif bölgeye bağlanması, diğer aktif bölgelerin substrat moleküllere olan afinitesini değiştirir. Pozitif kooperatiflik durumunda, ilk substratın bağlanması sonucu diğer substratlarına olan afinite (ilgi) artar. Negatif kooperatiflikte ise, ilk substratın bağlanması, diğer substratlara olan afinitenin azalmasına neden olur.
Allosterik enzimlere örnek olarak memeli tirozil tRNA-sentetaz (negatif kooperatiflik gösterir), bakteryel ve (pozitif kooperatiflik gösterir) sayılabilir.
Kooperatiflik oldukça yaygındır ve substrat konsantrasyonundaki değişikliğe bağlı olarak enzimlerin tepkilerinin düzenlemeye yarar. Pozitif kooperatiflik enzimleri [S]'ye daha duyarlı kılar ve dar bir konsantrasyon aralığında enzim aktivitesinin büyük değişklik göstermesini sağlar. Buna karşın, negatif kooperatiflik enzimleri [S]'de küçük değişkliklere az duyarlı kılar.
Michaelis-Menten kinetiğine uymayan sistemlerdeki kooperatiflik derecesini nicel olarak betimlemek için kullanılır. Türetilmiş Hill katsayısı n substratın bir aktif bölgeye bağlanmasının substratın diğer aktif bölgelere bağlanmasının ne kadar etkilediğinin ölçütüdür. Hill kasayısı <1 olması negatik kooperatiflik, Hill katsayısı >1 olması pozitif belirtir.
Sabit hâl öncesi kinetik

Bir enzim substratı ile karıştırlmasından sonraki ilk anda henüz bir ürün oluşmamış ve yoktur. Bunu takip eden milisaniylerin araştırması sabit hâl öncesi kinetik veya çoğuşma kinetiği (İngilizce Burst kinetics) olarak adlandırılır. Dolayısıyla, sabit hâl öncesi kinetiği, ulaşılana dek enzim substrat araürünlerinin (ES veya E* gibi) oluşumu ve tükenişi ile ilgilidir.
Bu yaklaşım ilk defa tarafından katalizlenen hidroliz reaksiyonuna uygulanmıştır. Genelde, bir araürünün tespiti, bir enzimin takip ettiği mekanizmayı araştırmada çok önemli bir delildir. Örneğin, yukarıda gbelirtilen pinpon mekanizmasında, hızlı kinetik ölçümler bir P ürününün enzimden salınmasını ve değişime uğramış bir E* enzim araürününün oluşumu izlenebilir. Kimotripsin durumunda, aktif bölgedeki nükleofilik serin substrata saldırarak bir asil-enzim araürünü meydana gelir.
Sağdaki resimde, reaksiyonun ilk birkaç saniyesi sırasında enzim hızla E* oluşturur. Sonra, sabit hâle yaklaştıkça reaksiyon yavaşlar. Reaksiyonun bu hızlı "çoğuşma" evresi, enzimdeki tek bir devinime karşılık gelir. Dolayısıyla, bu patlama sırasında salınan ürün miktarı (grafikte y eksenindeki kesim noktasına karşılık gelir), aynı zamanda reaksiyondaki fonksiyonel enzim miktarına eşittir.
Kimyasal mekanizma
Enzim kinetiğinin ölçümünde önemli bir amaç, enzim reaksiyonunun kimyasal mekanizmasının, yani substratın ürüne dönüştürülmesindeki kimyasal adımlar dizisinin belirlenmesidir. Yukarıda anlatılan bu kinetik yaklaşım ile araürünlerin ne hızla oluştuğu ve birbirine dönüştüğünü gösterir ama bu ara ürünlerin tam olarak ne olduğunu tespit edemez.
Farklı çözelti şartlarında yapılan veya biraz modifiye edilmiş enzim veya substratlar üzerinde yapılan kinetik ölçümler çoğu zaman bu kimyasal mekanizmanın anlaşılmasına yardımcı olur, çünkü hız belirleyici adım veya reaksiyon araürünlerini ortaya çıkarır. Örneğin, bir hidrojen atomuna olan bir kovalent bağın kırılması sık görülen bir hız belirleyici adımdır. Olasıl hidrojen transferlerinden hangisinin hız belirleyici olduğu, her bir hidrojeni (onun kararlı izotopu olan) döteryum ile değiştirmenin kinetik etkilerini ölçülerek gösterilebilir. dolayı döteryum bağlarının kırılması hidrojene olan kovalent bağların kırılmasından daha zordur, bu yüzden kritik hidrojen değiştirildiğinde reaksiyon hızı değişir. Başka izotop substitusyonları ile de (13C/12C ve 18O/16O) benzer etkilerin ölçülmesi mümkündür, ama bu etkiler aynı derecede bariz değildir.
İzotoplar substrat molekülünün farklı kısımlarının kaderini ortaya çıkarmak için de kullanılabilir. Örneğin, bazen nihai üründeki bir oksijenin kaynağını ayırt etmek zor olabilir, çünkü bu atom sudan veya substrattan geliyor olabilir. Hangisi olduğunu belirlemek için, reaksiyona katılan çeşitli moleküllere oksijenin kararlı izotopu olan 18O sistematik olarak substitüe edilir ve üründe bu izotopun varlığına bakılır. Kimyasal mekanizmanın anlaşılması için kullanılabilen diğer yöntemler, kinetik ve izotop etkileri farklı pH şartlarında, enzime bağlanan metal iyonları veya diğer kofaktörleri değiştirerek, korunmuş amino asit kalıntılarının ile veya substrat analoglarının varlığında enzimin davranışını incelemektir.
Enzim inhibisyonu ve aktivasyonu

Enzim inhibitörleri enzim aktivitesini azaltan veya ortadan kaldıran mpoleküllerdir, buna karşın enzim aktivatörleri enzimlerin katalitik aktivitesini artıran moleküllerdir. Bu etkileşimler ya tersinir (yani, inhibitörünün giderilmesi enzim aktivitesini geri getirir) veya tersinmez olabilir (yani inhibitör temelli olarak enzimi inaktive eder).
Tersinir inhibitörler
Geleneksel olarak, tersinir enzim inhibitörleri Km ve Vmax üzerindeki etkilerine bağlı olarak yarışmalı (İngilizce competitive), sınırlı yarışmalı (non-competitive), yarışmasız (uncompetitive) veya karışık inhibisyon olarak sınıflandırılmışlardır. Bu farklı etkiler inhibitörün ya E enzimine, ya ES enzim-substrat kompleksine ya da her ikisine birden bağlanmasının sonucudur, sağdaki şekil ve aşağıdaki tabloda gösterildiği üzere. İnhibitörün tipine ayırt etmek için, inhibitör konsantrasyonuna bağlı olarak enzim kinetiği incelenir. Bu dört tip inhibitör, inhibitör konsantrasyonuna bağlı olarak farklı ve grafikleri oluşturur. Sadelik için ilgili denklemlerde şu iki sembol kullanılır:
and
Burada Ki ve K'i, sırasıyla, enzime ve enzim-substrat kompleksinin ayrışma sabitleridir. Tersinir bir inhibitörün varlığında aşağıdaki tipik durumlar için gösterildiği üzere enzimin görünen Km ve Vmax değerleri, sırasıyla, (α/α')Km ve (1/α')Vmax olur.
| İnhibisyon tipi | Görünür Km | Görünür Vmax | ||
| Ki sadece | (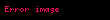 ) ) | yarışmalı |  |  |
| Ki' sadece | ( ) ) | yarışmasız | 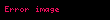 |  |
| Ki = Ki' | ( ) ) | sınırlı yarışmalı | 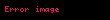 |  |
| Ki ≠ Ki' | ( ) ) | karışık | 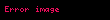 |  |
ile enzim kinetik verilerini hız denklemlerine uydurunca ayrışma katsayıları Ki ve K'i için yüksek doğruluklu olarak kestirmek mümkün olur.
Denklemlerin basit bir düzenlemesi inhibitör bağlanması ve maksimum hız arasındaki ilişkiyi ortaya koyar.
Aşağıya sıfır ekleyince ([I]-[I])
[I]+Ki ile bölünce
Yani, reaksiyon hızının substrat ile etkileşen enzim oranına bağlı olduğu Michaelis–Menten denklemine benzer şekilde,
substrata bağlı enzim oranı
inhibitöre bağlı enzim oranı
İnhibitörün etkisi, inhibitörle etkileşen enzim popülasyonunun yüzdesinin bir sonucudur. Bu denklemle ilgili tek sorun, inhbitör bağlanması ile enzimin mutlak olarak inhibe olduğunu varsaymasıdır, oysa bu etki substrat kullanımının %100 ila %1'in biraz fazlası arasında değişebilir. Değişken dereceli inhibisyonu hesaba katmak için denkleme bir delta Vmax terimi eklenir.
veya
Bu terim sayesinde, inhibitör popülasyondaki enzim molekülleri ile etkileşirken kalan enzim aktivitesi tanımlananbilir. Ayrıca, bu terimin denkleme dahil edilmesinin bir avantajı, ikincil Vmax terimi ilk terimden daha büyük olması halinde aktivasyon olasılığının da hesaplanmasını mümkün kılmasıdır. Aktivasyon olasılığını hesaba katmak için, İnhibitör için kullanılan "I" harfi yerine, aşağıda "X" olarak gösterilen bir modifikatör kavramına geçilebilir.
Bu terminoloji Michaelis-Menten denklemineki maksimal hız ile ilşkili kinetik etkileri daha basit bir şekilde ele almaya sağlamasına karşın, Km ile ilişkili etkileri tarif etmekte ortaya çıkabilen potansiyel problemleri de ortaya çıkarır. Enzimin substratına afinitesi ile ilgili olan Km, çoğu durumda enzim-inhbitör etkileşiminin sonucu olarak enzimin bağlanma yerindeki potansiyel değişikliklerle ilişkili olmalıdır. Bu yüzden, Vmax'yi modüle eden, yukarıdaki benzer bir terim olması çoğu durum için uygun olurdu: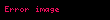
Tersinmez inhibitörler
Enzim inhibitörleri enzimleri tersinmez olarak da inaktive edebilirler, genelde aktif bölgedeki amino asit kalıntısını değişime uğratarak. Kendilerini de yok ettikeri için "intihar substratı" olarak düşünülebilecek olan bu inhibitörler, reaksiyon sırasında üssel bir azalma gösterirler ve konsantrasyona bağlı olarak enzime olan etkileri genelde doyum özelliği gösterir. Doyum noktasının altında bu reaksiyonlar inhibitör için birinci derece reaksiyon kinetiği gösterirler.
Kataliz mekanizması

Enzim-substrat etkileşimi ile ilgili favori model, indüklenmiş uyum modelidir. Bu modele göre, enzim ile substrat arasındaki ilk etkileşimin zayıftır, ancak bu zayıf etkileşimler enzimde konformasyonel değişiklikler doğurur (indükler) ve bunun sonucu olarak bağlanma güçlenir. Bu konformasyonel değişiklikler ayrıca aktif bölgedeki katalitik amino asit kalıntılarını, substratta reaksiyon sonucu değişecek kimyasal bağların yakınına getirir. Konformasyonel değişimler veya (İng. dual polarisation interferometry) ile ölçülebilir. Bağlanma olduktan sonra, kataliz mekanizmaları sayesinde, reaksiyon için alternatif kimyasal yolaklar sağlanarak reaksiyonun geçiş halinin enerjisi alçalır. Bunun olmasını sağlayan kataliz mekanizmaları arasında: bağ gerginliği yoluyla kataliz; yakınlık ve doğrultu ile kataliz; aktif bölge proton vericileri veya alıcıları ile kataliz; kovalent kataliz; ve kuvantum tünellemesi sayılabilir.
Enzim kinetiği ölçümleri bir enzim tarafından hangi kataliz yolunun kullanıldığını kanıtlayamaz. Ancak, bazı kinetik veriler, başka yöntemlerle doğrulanabilecek olasılıkları öne sürebilir. Örneğin, sabit-hâl öncesi çoğuşmalı pinpon kinetiği gösteren bir reaksiyon, kovalent katalizin bu enzim reaksiyonunda önemli olabileceğini ima eder. Alternatif olarak, Vmax üzerinde kuvvetli bir pH etkisi olması ama Km üzerinde bir pH etksisi olmaması, katalizin olabilmesi için aktif bölgedeki bir amino asit kalıntısının belli bir iyonizasyon hâlinde olması gerektiğine işaret edebilir.
Ayrıca bakınız
Dipnotlar
α. ^
β. ^
γ. ^
δ. ^
Kaynakça
- ^ {{Kitap kaynağı|yazar=Wrighton, Mark S.; Ebbing, Darrell D. |başlık=General chemistry |yayıncı=Houghton Mifflin |yer=Boston |yıl=1993 |isbn=0-395-63696-5 |basım=4.4
- ^ Danson, Michael; Eisenthal, Robert (2002). Enzyme assays: a practical approach. Oxford [Oxfordshire]: Oxford University Press. ISBN .
- ^ Xie XS, Lu HP (Haziran 1999). . J. Biol. Chem. 274 (23). ss. 15967-70. doi:10.1074/jbc.274.23.15967. (PMID) 10347141. 31 Mayıs 2009 tarihinde kaynağından arşivlendi. Erişim tarihi: 13 Temmuz 2011.
- ^ Lu H (2004). "Single-molecule spectroscopy studies of conformational change dynamics in enzymatic reactions". Current pharmaceutical biotechnology. 5 (3). ss. 261-9. doi:10.2174/1389201043376887. (PMID) 15180547.
- ^ Schnell J, Dyson H, Wright P (2004). "Structure, dynamics, and catalytic function of dihydrofolate reductase". Annual review of biophysics and biomolecular structure. Cilt 33. ss. 119-40. doi:10.1146/annurev.biophys.33.110502.133613. (PMID) 15139807.
- ^ Gibson QH (1969). "Rapid mixing: Stopped flow". Methods Enzymol. Cilt 16. ss. 187-228. doi:10.1016/S0076-6879(69)16009-7.
- ^ Duggleby RG (1995). "Analysis of enzyme progress curves by non-linear regression". Methods Enzymol. Cilt 249. ss. 61-90. doi:10.1016/0076-6879(95)49031-0. (PMID) 7791628.
- ^ Murray JB, Dunham CM, Scott WG (Ocak 2002). "A pH-dependent conformational change, rather than the chemical step, appears to be rate-limiting in the hammerhead ribozyme cleavage reaction". J. Mol. Biol. 315 (2). ss. 121-30. doi:10.1006/jmbi.2001.5145. (PMID) 11779233.
- ^ Michaelis L. and Menten M.L. Kinetik der Invertinwirkung Biochem. Z. 1913; 49:333–369 İngilizce çevirisi 9 Mayıs 2008 tarihinde Wayback Machine sitesinde . Accessed 6 April 2007
- ^ Stroppolo ME, Falconi M, Caccuri AM, Desideri A (2001). "Superefficient enzymes". Cell. Mol. Life Sci. 58 (10). ss. 1451-60. doi:10.1007/PL00000788. (PMID) 11693526.
- ^ Walsh R, Martin E, Darvesh S. A method to describe enzyme-catalyzed reactions by combining steady state and time course enzyme kinetic parameters. Biochim Biophys Acta. 2010 Jan;1800:1-5
- ^ Jones ME (1 Ocak 1992). "Analysis of algebraic weighted least-squares estimators for enzyme parameters". Biochem. J. 288 (Pt 2). ss. 533-8. (PMC) 1132043 $2. (PMID) 1463456.
- ^ a b Tseng SJ, Hsu JP (Ağustos 1990). "A comparison of the parameter estimating procedures for the Michaelis-Menten model". J. Theor. Biol. 145 (4). ss. 457-64. doi:10.1016/S0022-5193(05)80481-3. (PMID) 2246896.
- ^ Bravo IG, Busto F, De Arriaga D; ve diğerleri. (Eylül 2001). "A normalized plot as a novel and time-saving tool in complex enzyme kinetic analysis". Biochem. J. 358 (Pt 3). ss. 573-83. (PMC) 1222113 $2. (PMID) 11577687.
- ^ Almaas E, Kovács B, Vicsek T, Oltvai ZN, Barabási AL (Şubat 2004). "Global organization of metabolic fluxes in the bacterium Escherichia coli". Nature. 427 (6977). ss. 839-43. doi:10.1038/nature02289. (PMID) 14985762.
- ^ Reed JL, Vo TD, Schilling CH, Palsson BO (2003). "An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR)". Genome Biology. 4 (9). ss. R54. doi:10.1186/gb-2003-4-9-r54. (PMC) 193654 $2. (PMID) 12952533.
- ^ Denklemin türetilmesi için bakınız: here
- ^ Dirr H, Reinemer P, Huber R (Mart 1994). "X-ray crystal structures of cytosolic glutathione S-transferases. Implications for protein architecture, substrate recognition and catalytic function". Eur. J. Biochem. 220 (3). ss. 645-61. doi:10.1111/j.1432-1033.1994.tb18666.x. (PMID) 8143720.
- ^ Stone SR, Morrison JF (Temmuz 1988). "Dihydrofolate reductase from Escherichia coli: the kinetic mechanism with NADPH and reduced acetylpyridine adenine dinucleotide phosphate as substrates". Biochemistry. 27 (15). ss. 5493-9. doi:10.1021/bi00415a016. (PMID) 3052577.
- ^ Fisher PA (1994). "Enzymologic mechanism of replicative DNA polymerases in higher eukaryotes". Prog. Nucleic Acid Res. Mol. Biol. Cilt 47. ss. 371-97. doi:10.1016/S0079-6603(08)60257-3. (PMID) 8016325.
- ^ Akerman SE, Müller S (Ağustos 2003). "2-Cys peroxiredoxin PfTrx-Px1 is involved in the antioxidant defence of Plasmodium falciparum". Mol. Biochem. Parasitol. 130 (2). ss. 75-81. doi:10.1016/S0166-6851(03)00161-0. (PMID) 12946843. 20 Şubat 2008 tarihinde kaynağından . Erişim tarihi: 13 Temmuz 2011.
- ^ Bravo IG, Barrallo S, Ferrero MA, Rodríguez-Aparicio LB, Martínez-Blanco H, Reglero A (Eylül 2001). "Kinetic properties of the acylneuraminate cytidylyltransferase from Pasteurella haemolytica A2". Biochem. J. 358 (Pt 3). ss. 585-98. (PMC) 1222114 $2. (PMID) 11577688.
- ^ Kraut J (1977). "Serine proteases: structure and mechanism of catalysis". Annu. Rev. Biochem. Cilt 46. ss. 331-58. doi:10.1146/annurev.bi.46.070177.001555. (PMID) 332063.[]
- ^ Ricard J, Cornish-Bowden A (Temmuz 1987). "Co-operative and allosteric enzymes: 20 years on". Eur. J. Biochem. 166 (2). ss. 255-72. doi:10.1111/j.1432-1033.1987.tb13510.x. (PMID) 3301336.
- ^ Ward WH, Fersht AR (Temmuz 1988). "Tyrosyl-tRNA synthetase acts as an asymmetric dimer in charging tRNA. A rationale for half-of-the-sites activity". Biochemistry. 27 (15). ss. 5525-30. doi:10.1021/bi00415a021. (PMID) 3179266.
- ^ Helmstaedt K, Krappmann S, Braus GH (Eylül 2001). "Allosteric regulation of catalytic activity: Escherichia coli aspartate transcarbamoylase versus yeast chorismate mutase". Microbiol. Mol. Biol. Rev. 65 (3). ss. 404-21, table of contents. doi:10.1128/MMBR.65.3.404-421.2001. (PMC) 99034 $2. (PMID) 11528003. 11 Haziran 2011 tarihinde kaynağından . Erişim tarihi: 13 Temmuz 2011.
- ^ Schirmer T, Evans PR (Ocak 1990). "Structural basis of the allosteric behaviour of phosphofructokinase". Nature. 343 (6254). ss. 140-5. doi:10.1038/343140a0. (PMID) 2136935.
- ^ Hill, A. V. The possible effects of the aggregation of the molecules of haemoglobin on its dissociation curves. J. Physiol. (Lond.), 1910 40, iv-vii.
- ^ Hartley BS, Kilby BA (Şubat 1954). "The reaction of p-nitrophenyl esters with chymotrypsin and insulin". Biochem. J. 56 (2). ss. 288-97. (PMC) 1269615 $2. (PMID) 13140189.
- ^ a b Fersht, Alan (1999). Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding. San Francisco: W.H. Freeman. ISBN .
- ^ Bender ML, Begué-Cantón ML, Blakeley RL; ve diğerleri. (Aralık 1966). "The determination of the concentration of hydrolytic enzyme solutions: alpha-chymotrypsin, trypsin, papain, elastase, subtilisin, and acetylcholinesterase". J. Am. Chem. Soc. 88 (24). ss. 5890-913. doi:10.1021/ja00976a034. (PMID) 5980876.
- ^ Cleland WW (Ocak 2005). "The use of isotope effects to determine enzyme mechanisms". Arch. Biochem. Biophys. 433 (1). ss. 2-12. doi:10.1016/j.abb.2004.08.027. (PMID) 15581561. 20 Şubat 2008 tarihinde kaynağından . Erişim tarihi: 5 Eylül 2008.
- ^ Northrop D (1981). "The expression of isotope effects on enzyme-catalyzed reactions". Annu. Rev. Biochem. Cilt 50. ss. 103-31. doi:10.1146/annurev.bi.50.070181.000535. (PMID) 7023356.
- ^ Baillie T, Rettenmeier A (1986). "Drug biotransformation: mechanistic studies with stable isotopes". Journal of clinical pharmacology. 26 (6). ss. 448-51. (PMID) 3734135.
- ^ Cleland WW (1982). "Use of isotope effects to elucidate enzyme mechanisms". CRC Crit. Rev. Biochem. 13 (4). ss. 385-428. doi:10.3109/10409238209108715. (PMID) 6759038.
- ^ Christianson DW, Cox JD (1999). "Catalysis by metal-activated hydroxide in zinc and manganese metalloenzymes". Annu. Rev. Biochem. Cilt 68. ss. 33-57. doi:10.1146/annurev.biochem.68.1.33. (PMID) 10872443.
- ^ Kraut D, Carroll K, Herschlag D (2003). "Challenges in enzyme mechanism and energetics". Annu. Rev. Biochem. Cilt 72. ss. 517-71. doi:10.1146/annurev.biochem.72.121801.161617. (PMID) 12704087.
- ^ Leatherbarrow RJ (Aralık 1990). "Using linear and non-linear regression to fit biochemical data". Trends Biochem. Sci. 15 (12). ss. 455-8. doi:10.1016/0968-0004(90)90295-M. (PMID) 2077683.
- ^ Walsh R, Martin E, Darvesh S. A versatile equation to describe reversible enzyme inhibition and activation kinetics: modeling beta-galactosidase and butyrylcholinesterase. Biochim Biophys Acta. 2007 1770:733-46.
- ^ Koshland DE (Şubat 1958). "Application of a Theory of Enzyme Specificity to Protein Synthesis". Proc. Natl. Acad. Sci. U.S.A. 44 (2). ss. 98-104. doi:10.1073/pnas.44.2.98. (PMC) 335371 $2. (PMID) 16590179.
- ^ Hammes G (2002). "Multiple conformational changes in enzyme catalysis". Biochemistry. 41 (26). ss. 8221-8. doi:10.1021/bi0260839. (PMID) 12081470.
- ^ Sutcliffe M, Scrutton N (2002). "A new conceptual framework for enzyme catalysis. Hydrogen tunnelling coupled to enzyme dynamics in flavoprotein and quinoprotein enzymes". Eur. J. Biochem. 269 (13). ss. 3096-102. doi:10.1046/j.1432-1033.2002.03020.x. (PMID) 12084049. 8 Kasım 2006 tarihinde kaynağından . Erişim tarihi: 13 Temmuz 2011.
Konuyla ilgili yayınlar
Giriş düzeyi
- {{Kitap kaynağı|yazar=Cornish-Bowden, Athel |başlık=Fundamentals of enzyme kinetics |yayıncı=Portland Press |yer=Londra|yıl=2004 |isbn=1-85578-158-1 |basım=3.3 (İngilizce)
- Stevens, Lewis; Price, Nicholas C. (1999). Fundamentals of enzymology: the cell and molecular biology of catalytic proteins. Oxford [Oxfordshire]: Oxford University Press. ISBN . (İngilizce)
- Bugg, Tim (2004). Introduction to Enzyme and Coenzyme Chemistry. Cambridge, MA: Blackwell Publishers. ISBN . (İngilizce)
İleri düzey
- Segel, Irwin H. (1993). Enzyme kinetics: behavior and analysis of rapid equilibrium and steady state enzyme systems (New bas.). New York: Wiley. ISBN . (İngilizce)
- Fersht, Alan (1999). Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding. San Francisco: W.H. Freeman. ISBN . (İngilizce)
- Santiago Schnell, Philip K. Maini (2004). (PDF). Comments on Theoretical Biology. Cilt 8. ss. 169-87. doi:10.1080/08948550302453. 21 Şubat 2006 tarihinde kaynağından (PDF) arşivlendi. Erişim tarihi: 13 Temmuz 2011. (İngilizce)
- Walsh, Christopher (1979). Enzymatic reaction mechanisms. San Francisco: W. H. Freeman. ISBN . (İngilizce)
- Cleland, William Wallace; Cook, Paul (2007). Enzyme kinetics and mechanism. New York: Garland Science. ISBN . (İngilizce)
Dış bağlantılar
- Bir enzim ölçümünün animasyonu 6 Ekim 2011 tarihinde Wayback Machine sitesinde . — Shows effects of manipulating assay conditions.
- MACiE 8 Ağustos 2011 tarihinde Wayback Machine sitesinde . — Enzim mekanizmaları veritabanı.
- ENZYME6 Ekim 2006 tarihinde Wayback Machine sitesinde . — Expasy enzim adlandırma sistemi veritabanı.
- — Enzim katalitik mekanizmaları veritabanı.
- BRENDA4 Kasım 2011 tarihinde Wayback Machine sitesinde . — Kapsamlı bir enzim veritabanı, substratlar, inhibitörler ve reaksiyon çizimleri bulunmaktadır.
- — Reaksiyon kinetkileri veritabanı.
- — Birkaç enzim reaksiyonun mekanizmasının animasyonu.
- — Enzim kinetiğindeki kavramlar ve terminolojinin kapsamlı bir açıklaması.
- — Enzim kinetiği hakkında çevrimiçi, kolay anlaşılır bir eğitsel.
- Enzim kinetiği animasyonlu eğitsel 24 Temmuz 2011 tarihinde Wayback Machine sitesinde .
wikipedia, wiki, viki, vikipedia, oku, kitap, kütüphane, kütübhane, ara, ara bul, bul, herşey, ne arasanız burada,hikayeler, makale, kitaplar, öğren, wiki, bilgi, tarih, yukle, izle, telefon için, turk, türk, türkçe, turkce, nasıl yapılır, ne demek, nasıl, yapmak, yapılır, indir, ücretsiz, ücretsiz indir, bedava, bedava indir, mp3, video, mp4, 3gp, jpg, jpeg, gif, png, resim, müzik, şarkı, film, film, oyun, oyunlar, mobil, cep telefonu, telefon, android, ios, apple, samsung, iphone, xiomi, xiaomi, redmi, honor, oppo, nokia, sonya, mi, pc, web, computer, bilgisayar
Enzim kinetigi enzimler tarafindan katalizlenen kimyasal reaksiyonlarin bilimidir Enzim kinetiginde reaksiyon hizi olculur ve reaksiyon sartlarini degistirmenin etkisi arastirilir Bir enzimin kinetiginin bu sekilde calisilmasi enzimin katalitik mekanizmasini metabolizmadaki rolunu aktivitesinin nasil kontrol edildigini ve bir ilac veya zehrin enzimi nasil edebilecegini ortaya koyabilir enzimi aktif bolgeye bagli iki substrati ile sagda ve NADPH solda Protein ile gosterilmistir alfa sarmallar kirmizi beta yapraklar sari ve donmeler mavidir 7DFR 2 Mart 2012 tarihinde Wayback Machine sitesinde koordinatlarlarindan uretilmistir Enzimler genelde diger molekulleri enzimin substratlari manipule eden protein molekulleridir Bu hedef molekuller enzimin aktif bolgesine baglanir ve enzim mekanizmasi denen bir seri adimlar sonucunda urunlere donusur Bu mekanizmalar tek substratli ve cok substratli mekanizmalar olarak ayrilabilir Tek bir substrata baglanan enzimler gibi uzerinde yapilan kinetik calismalar enzimin substratina baglanma ilgisini ve devir hizini olcmeyi amaclar Enzimler birden cok substrata baglaninca sagda gosterilen gibi enzim kinetigi bu substratlarin baglanma sirasini ve urunlerin salinma sirasini gosterebilir Bir tek substrata baglanip birden cok urun salan enzimlere ornek proteazlardir bunlar bir protein substrati kesip iki polipeptit urun olustururlar Baska enzimler iki substrati birlestirir orenegin DNA polimeraz in DNA ya bir nukleotit eklemesi gibi Bu mekanizmalar genelde karmasik bir adimlar dizisi olsa da tipik olarak bir hiz belirleyici basamak tum reaksiyonun hizini belirler Bu kimyasal bir reaksiyon veya enzim veya substratlarinda konformasyonel bir degisim olabilir urunlerin enzimden salinmasi sirasinda goruldugu gibi Enzimin yapisinin bilinmesi kinetik verilerin yorumlanmasinda faydalidir Ornegin yapinin bilinmesi sayesinde substrat ve urunlerin kataliz sirasinda nasil baglandiklari reaksiyon sirasindan hangi degisikliklerin meydana geldigi ve hatta mekanizmadaki belli amino asit kalintilarinini rolleri tahmin edilebilir Bazi enzimler mekanizma sirasinda onemli derecede bicimlerini degistirirler boylesi durumlarda enzimin tek basina yapisinin ve enzimatik reaksiyona girmeyen substrat analoglari ona bagli ikenki yapisinin belirlenmesi yararli olur Tum biyolojik katalizorler protein degildir ribozim ve ribozom gibi RNA temelli katalizorler pek cok hucresel islev icin esastirlar ornegin RNA ucbirlestirmesi splicing ve ceviri translasyon gibi Ribozimler ve enzimler arasindaki temel fark RNA katalizorlerin nukleotitlerden enzimlerin ise amino asitlerden olusmasidir Ribozimler daha sinirli bir reaksiyonlar grubunu katalizler ama reaksiyon mekanizmalari ve kinetikleri protein enzimleri ile ayni yontemlerle analiz edilebilir ve siniflandirilabilir Genel ilkelerSubstrat konsantrasyonu arttikca reaksiyon hizi da artar ama yuksek substrat konsantrasyonunda doyuma ulasir Bir enzim tarafindan katalizlenen reaksiyon katalizlenmeyen reaksiyon ile ayni denge ozelliklerine sahiptir Diger katalizorler gibi enzimler substrat ve urunler arasindaki dengenin konumunu degistirmezler Ancak katalizlenmeyen bir kimyasal reaksiyondan farkli olarak enzimle katalizlenen reaksiyonlar doyum kinetigi gosterirler Belli bir enzim konsantrasyonu ve nispeten dusuk substrat konsantrasyonu icin reaksiyon hizi substrat konsantrasyonu ile dogrusal orantili olarak artar enzim molekullerinin cogu reaksiyonu katalizlemek icin musaittir ve substrat konsantrasyonun artirilmasi enzim ve substrat molekullerinin birbirine rastlama hizinin artmasi demektir Ancak nispeten yuksek substrat konsantrasyonlarida reasiyon hizi asimptotik sekilde teorik maksimuma yaklasir enzim aktif bolgelerinin hemen hepsi doludur ve reaksiyon hizini enzimin kendine has icsel devir hizi belirler Bu iki sinir durumun ortasindaki substrat konsantrasyonu KM olarak belirtilir Bir diger deyisle en yuksek reaksiyon hizinin Vmax yarisinin elde edildigi substrat konsantrasyonu KM olarak tanimlanir Bir enzimin en onemli iki kinetik ozelligi enzimin belli bir substrat ile ne kadar cabuk doyuma ulastigi ve ulasabildigi en hizli reaksiyon hizidir Bu ozelliklerin bilinmesi bir enzimin hucre icinde ne yaptigini ve o sartlardaki degisiklere nasil tepki verdigi hakkinda fikir verir Enzim olcumleriBir enzim reaksiyonu icin ilerleme egrisi Ilk bastaki egim reaksiyonun ilk hizi v dir Michaelis Menten denklemi bu egimin substrat konsantrasyonu ile nasil degistigini betimler Enzim olcumleri enzim reakasiyonunun hizini olcen laboratuvar yontemleridir Enzimler katalizledikleri reaksiyonlar tarafindan tuketilmedigi icin enzim olcumlerinde reaksiyon hizini takip etmek icin genelde ya substrat konsantrasyonu ya da urun konsantrasyonu izlenir Olcum icin pek cok yontem vardir olcumlerde substrat ve urunlerdeki isik degisim gozlemlenir radyometrik olcumlerde radyoaktivitenin bir urune dahil olusu veya substrattan ayrilmasina bakarak zamana gore urun olusumunu olculur Spektrofotometrik olcumler en kullanislidir cunku reaksiyon hizinin surekli olarak olculmesi mumkundur Radyometrik olcumler numunelerin alinip onlarin olcumunu gerektirse de yani kesintili olcumlerdir genelde daha duyarlidirlar ve cok dusuk enzim aktivite duzeyleri olculebilir Benzer bir yaklasim kutle spektrometresi kullanarak urune stabil bir izotopun enkorpore olmasinin veya substrattan salinmasinin izlenmesidir En duyarli enzim olcumleri bir mikroskop icinden odaklanmis bir lazer kullanarak reaksiyonunu katalizleyen tek bir enzim molekulunu gozlemler Bu olcumler enzimin reaksiyon mekanizmasi sirasinda kofaktorlerin floresansindaki degisimi veya protein uzerinde belli konumlara baglanmis degisimi olcer kataliz sirasindaki hareketleri izlemek icin Milyonlarca enzim molekulunun ortalama davranisini gozlemleyen geleneksel enzim kinetigine karsin bu tur calismalar tekil enzimlerin kinetigi ve dinamigi hakkinda yeni bir bakis acisi saglar Bir enzim olcumu icin bir ilerleme egrisi ornegi yukarida gosterilmistir Enzim reaksiyonun baslamasinda sonra kisa bir sure icin dogrusal sekilde urun meydana getirir Reaksiyon ilerledikce ve substrat tuketildikce hizi gittikce azalir substrat doyum seviyesinde olmadikca Ilk ve en yuksek hizi olcmek icin enzim olcumleri tipik olarak substratin sadece yuzde birkaci tuketilene kadar yapilir Bu ilk hiz doneminin uzunlugu olcum sartlarina baglidir ve milisaniyeleden saatlere kadar degisebilir Ancak sivilarin cok hizli karsmasini saglayan araclar bir saniyeden kisa surelerde ilk hizlarin olculebilmesini olanak verir Asagida anlatildigi uzere bu cok hizli olcumler sabit durum oncesi pre steady state kinetiginin olculmesi icin sarttir Cogu enzim kinetik calismalari enzim reaksiyonlarinin bu ilk yaklasik dogrusal kismi uzerine yogunlasir Ancak tum reaksiyonu olcup bu verileri dogrusal olmayan bir uydurmak da mumkundur Enzim reaksiyonlarinin bu sekilde olculmesi ilerleme egrisi analizi Ing progress curve analysis olarak adlandirilir Eger ilk hiz ile dogru olculemeyecek kadar yuksek ise bu yaklasim yararli bir alternatiftir Tek substrat reaksiyonlariTek substrat mekanizmali enzimlere ornek olarak izomerazlar veya gibi gibi hucre ici liyazlar ve bir olan sayilabilir Ancak tek bir substrati olan bazi enzimler bu mekanizma kategorisi icine girmezler Bunun bir ornegi olan katalaz substrati olan hidrojen peroksit ile reaksiyona girince yukseltgenir okside olur ve sonra ikinci bir substrat molekulu tarafindan indirgenir Substrat tek olsa da modifiye olmus bir enzim ara urununun varligi katalaz mekanizmasinin aslinda bir pinpon masa tenisi mekanizmasi oldugunu gosterir Bu mekanizma tipi asagida Cok substratli reaksiyonlar bolumu altinda aciklanmistir Michaelis Menten kinetigi Substrat konsantrasyonu ile hiz arasindaki iliskiyi gosteren bir enzim doyum egrisi Bir enzim reaksiyonu icin tek sustrat mekanizmasi k1 k 1 ve k2 ayri basamaklarin hiz sabitleridir Enzimle katalizlenmis reaksiyonlar doyumlu oldugu icin kataliz hizlari artan substrata dogrusal bir tepki gostermez Eger ilk reaksiyon hizi cesitli substrat konsantrasyonlari S ile gosterilir icin olculurse reaksiyon hizi v S arttikca artar sagda gosterildigi gibi Ancak S yukseldikce enzim doygunlasir sature olur ve hiz enzimin en buyuk hizi olan Vmax a ulasir Tek substratli reaksiyon icin Michaelis Menten kinetik modeli sagda gosterilmistir Enzim E ve substrati S arasinda once bir iki molekullu reaksiyon olup bir enzim substrat reaksiyonu ES meydana gelir Tek molekullu reaksiyon ES kcatE P displaystyle ES overset k cat longrightarrow E P icin enzim reaksiyonu cok karmasik olabilirse de tipik olarak hiz belirleyici tek bir enzimatik adim vardir Bu sayede bu reaksiyon tek bir katalitik basamagi olan tek molekullu hiz sabiti gorunuste kcat imis gibi modellenebilir Eger reaksiyon yolu bir veya birkac araurun uzerinden gidiyorsa kcat birkac ogesel elementer hiz sabitinin fonsiyonu olacaktir Oysa en basit durum olan tek bir ogesel reaksiyonda yani araurun olmamasi halinde ogesel tek molekullu sabit k2 ye esit olacaktir Gorunur tek molekullu hiz sabiti kcatdevir hizi olarak da adlandirilir ve bu saniyedeki katalizlenen en yuksek enzim reaksiyon sayisina karsilik gelir Michaelis Menten denklemi baslangic reaksiyon hizi v0 nin substrata baglanma denge konumuna ve hiz sabiti k2 ye nasil bagli oldugunu betimler v0 Vmax S KM S displaystyle v 0 frac V max mbox S K M mbox S Michaelis Menten denklemi sabitler KM def k2 k 1k1 KDVmax def kcat E tot displaystyle begin aligned K M amp stackrel mathrm def frac k 2 k 1 k 1 approx K D V max amp stackrel mathrm def k cat E tot end aligned Bu Michaelis Menten denklemi cogu tek substratli enzim kinetiginin temelinde yatar Mekanizma hakkinda genel bir varsayim araurun veya urun inhibisyonu olmadigi ve veya olmadigidir Bu denklemin altinda yatan iki onemli varsayim daha vardir Birincisi sabit durumumsuluk varsayimi Ing quasi steady state assumption veya sahte sabit durum hipotezi olarak adlandirilir substrata bagli enzim konsantrasyonundaki ve dolayisiyla bagli olmayan enzimin konsantrasyonundaki de degisim hizinin urun ve substratin konsantrasyonlarindaki degismeden cok daha az oldugudur dolayisiyla kompleksteki degisimin zamana bagli degisiminin sifira esitlenebilecegidir d ES dt 0 displaystyle d ES dt overset 0 Ikinci varsayim ise toplam enzim konsantrayonunun zaman icinde degismedigi yani E tot E ES const displaystyle E text tot E ES overset text const Bu denklemlerin turetilmesinin ayrintilari burada bulunabilir Michaelis sabiti KM nin deneysel tanimi enzim reaksiyon hizinin Vmax in yarisi oldugu substrat konsantrasyonudur Michaelis Menten denkleminde S Km substitusyonu yapilarak bu tanim dogrulanabilir ayrica grafik olarak da gosterilebilir Eger hiz belirleyici enzimatik adim substrat ayrisma hizina k2 k 1 displaystyle k 2 ll k 1 kiyasla yavas ise Michaelis sabiti KM kabaca ES kompleksinin ayrisma katsayisi KD ye esittir Eger S displaystyle S KM displaystyle K M ye kiyasla kucukse S KM S S KM displaystyle S K M S approx S K M ve cok az ES kompleksi olusur yani E 0 E displaystyle E 0 approx E Dolayisiyla urun olusma hizi v0 kcatKM E S eger S KM displaystyle v 0 approx frac k cat K M E S qquad qquad text eger S ll K M Yani urun olusma hizi hem enzim konsantrasyonuna hem de substrat konsantrasyonuna baglidir psodo ikinci derece hiz sabiti k2 KM displaystyle k 2 K M olan iki molekullu reaksiyonunun denklemine benzer bu denklem Bu sabit katalitik verimliligin bir olcutudur En verimli enzimler 108 1010 1 1 araliginda k2 KM displaystyle k 2 K M degerlerine sahiptir Bu enzimler o kadar verimlidir ki bir substat molekulune her rastladiklarinda bir reaksiyon katalizlerler ve dolayisiyla verimliligin teorik ust sinirina difuzyon sinirina ulasmislardir Bu enzimler cogu zaman mukemmel enzim olarak adlandirilir Michaelis Menten denkleminin zaman bagimli kinetik analizi icin kullanimi Michaelis menten denklemi tarafindan ongorulen reaksiyon hizlarindan yararlanilarak zaman bagimli substrat yokolusu ve urun olusumu dogrudan modellenebilir Bunu yapmak icin birinci derece kimyasal kinetik denklemi icine Michaelis Menten denklemi dahil edilir Birinci derece kimayasal kinetigin tarifinde Euler sayisinin kullanimi ile ilgili problemin farkina varmak gerekir yani e k hesaplamalara sistematik bir hata sokan bir ayrik katsayidir Bu tek bir katsayi kullanilarak yeniden yazilabilir bu yeni katsayi her zaman araligindan sonraki kalan substrat miktarina karsilik gelir S S 0 1 k t displaystyle S S 0 1 k t S S 0 1 v S 0 t displaystyle S S 0 1 v S 0 t S S 0 1 Vmax S 0 KM S 0 S 0 t displaystyle S S 0 1 V max S 0 K M S 0 S 0 t Michaelis Menten denklemi icin dogrusal grafikler Kinetik veri icin Lineweaver Burk veya cifte reciprocal grafigi eksen kesim noktalarinin ve egimin anlamlari gosterilmistir Yukarida gosterilen S ye bagli v grafigi dogrusal degildir grafik dusuk S icin baslangicta dogrusal olsa dahi yuksek S de egilerek doyuma ulasir Bu konuda yapilan ilk calismalarda bu nonlineerlik KM ve Vmax in hatasiz olarak kestirilmesini zor kilmaktaydi Bu yuzden bazi arastirmacilar Michaelis Menten denkleminin lineerlestiren grafik yontemler gelistirdiler ve gibi Bu lineer gosterimlerin hepsi verinin goruntulenmesi icin yararlidir ama kinetik parametrelerinin belirlenmesi icin gunumuzde bilgisayar yazilimlari mevcuttur ve nonlineer regresyon yontemleri kullanarak daha hatasiz sonuclar elde edilmesi mumkundur veya cifte evrik Ing reciprocal grafigi kinetik veriyi goruntulemek icin yaygin kullanilir Bunu elde etemk icin Michaelis Menten denkleminin iki tarafinin yapilir Sagda gosterildigi gibi bu Michaelis Menten denkleminin dogrusal bir bicimidir ve y mx c denklemli bir dogru uretir y ekseni kesisim noktasi 1 Vmax ye x eksen kesisimi 1 KM ye karsilik gelir 1v KMVmax S 1Vmax displaystyle frac 1 v frac K M V max mbox S frac 1 V max Tabii ki negatif 1 S degerleri icin deneysel degerler elde edilemez 1 S 0 alt siniri y kesisimi sonsuz substrat konsantrasyonuna karsilik gelir orada 1 v 1 Vmax dir sagda gosterildigi gibi Dolayisiyla x kesisimi pozitif konsantrasyonlarda elde edilen deneysel verilerin bir ekstrapolasyonudur Daha genel olarak Lineweaver Burk grafigi dusuk konsantrasyonlarda elde edilen olcumlerin onemini carpitir ve dolayisiyla Vmax ve KM nin hatali kestirilmesine yol acabilir Daha hatasiz bir lineer grafikleme yontemi Bu durumda v v S ye bagli olarak grafiklenir Yaygin kullanilan yontemlerden ucuncusu olan S v S ye bagli olarak grafiklenir Genelde data normalizasyonu deneysel is miktarini azaltmaya yarar sonucun guvenilirligini artirabilir ve hem grafik hem de sayisal analiz icin uygundur Kinetik sabitlerin pratik anlami Enizm kinetiginin arastirilmasi iki temel nedenden dolayi onemlidir Birincisi enzimlerin nasil calistiginin anlasilmasini saglar ikincisi enzimlerin organizmalarda nasil davrandigini ongormeye yardimci olur Yukarida tanimlanan kinetik katsayilar KM ve Vmax metabolima kontrolunde enzimlerin nasil calistiginin anlasilmasinda onemli rol oynar Bu ongorulerin yapilmasi siradan degildir basit sistemler icin dahi Ornegin mitokondride bulunan tarafindan meydana gelir Oksaloasetat sonra veya tarafindan tuketilebilir ve sirasiyla sitrik asit dongusu veya aspartik asit biyosentezini besleyebilir Bu yolaklardan her birine ne kadar oksaloasetat gittigini ongormek icin oksaolasetat konsantrasyonunu bilmek ayrica bu enzimlerin konsantrasyon ve kinetik ozelliklerini bilmek gerekir Metabolik yolaklarin davranislarini ongorusunun en karmasik kismi tum bir organizmaya ait matematiksel modellerin icine girmesi gereken kinetik ve gen ifadesi verilerinin sentezini yapilmasindadir Buna alternatif olarak kinetik metabolizma probleminin faydali bir sadelestirmesi enzim kinetiklerini dikkate almayip sadece reaksiyon aginin stokyometresi hakkindaki bilgileri dayanmaktir bu yontem olarak adlandirilir Ararurunlu Micahelis Menten kinetigi Basit olan su durum ile baslayalim E S k1k 1ES k2EI k3E P displaystyle begin aligned E S underset k 1 overset k 1 begin smallmatrix displaystyle longrightarrow displaystyle longleftarrow end smallmatrix ES overset k 2 longrightarrow EI overset k 3 longrightarrow E P end aligned burada bir enzim ve araurun mevcuttur araurun ikinci adimda urune donusur Bu durumda cok benzer bir denklem ortaya cikar v0 kcat S E 0KM S displaystyle begin aligned v 0 amp k cat frac S E 0 K M prime S end aligned ama sabitler farklidir KM def k3k2 k3KM k3k2 k3 k2 k 1k1kcat def k3k2k2 k3 displaystyle begin aligned K M prime amp stackrel mathrm def frac k 3 k 2 k 3 K M frac k 3 k 2 k 3 cdot frac k 2 k 1 k 1 k cat amp stackrel mathrm def dfrac k 3 k 2 k 2 k 3 end aligned Sinirlayici durum olan k3 k2 displaystyle k 3 gg k 2 icin yani EI den E P ye olan adim bir evvelki adimdan cok daha hizli olunca ilk denklemi elde edilir Matematik olarak KM KM displaystyle K M prime approx K M ve kcat k2 displaystyle k cat approx k 2 Cok substratli reaksiyonlarCok substratli reaksiyonlarda subsratlarin nasil baglandigini ve hangi sirayla baglandigini belirten karmasik hiz denklemleri vardir Eger A substratinin konsantrasyonu sabit tutulur ve B substratininki degistirilirse bu reaksiyonlarin analizi cok basitlesir Bu sartlar altinda enzim tek substratli bir enzim gibi davranis gosterir v yi S ye bagli olarak grafiklendiginde B substati icin gorunur KM ve Vmax sabitleri elde edilir Eger bu olcumler A nin farkli sabit konsantrasyonlari icin yapilirsa bu veriler kullanilarak reaksiyonun mekanizmasi anlasilabilir Iki substrat A ve B yi alip bunlari iki urun P ve Q ya donusturen bir enzim icin iki tip mekanizma vardir uclu kompleks veya pinpon Uclu kompleks mekanizmalari Bir enzim reaksiyonu icin rastgele sirali uclu kompleks mekanizmasi Reaksiyon bir cizgi seklinde gosterilmistir substratlar A ve B veya urunler P ve Q yu iceren enzim araurunleri cizginin altina yazilmistir Bu enzimlerde her iki substrat enzime ayni anda baglanip bir EAB uclu kompleksi meydana getirir Baglanma sirasi rastgele olabilir rastgele mekanizmada veya substratlarin belli bir sira sira ile baglanmasi gerekebilir sirali mekanizmada Uclu kompleks mekanizmali bir enzim icin bir v S egriler kumesi sabit A degisen B bir Lineweaver Burk grafiginde cizilirse dogrular bir noktada kesisir Uclu kompleks mekanizmali enzimler arasinda and sayilabilir Dihidrofoloat reduktaz ve DNA polimeraz icin uclu kompleks mekanizmalarini gosteren kisa animasyonlar su baglantida izlenebilir Ref label B b none g Pinpon mekanizmasi Bir enzim reaksiyonu icin pinpon mekanizmasi Araurunler A ve B substratlari veya P ve Q urunlerini icerir Sagda gosterildigi gibi pinpon mekanizmali enzimler iki durumda bulunabilir asil halleri olan E ve kimyasal olarak modifiye bir hal olan E Enzimin bu modifiye olmus hali olarak adlandirilir Bu mekanizmada substrat A enzime baglanir aktif bolgeye bir kimyasal grup aktararak enzimi E a donusturur ve sonra enzimden salinir Ilk substrat salindiktan sonra ancak substrat B baglanabilir ve modifiye olmus enzimle reaksiyona girip modifiye olmamis E bicimini yeniden olusturur Pinpon mekanizmali bir enzim icin bir v S egriler kumesi sabit A degisen B Lineweaver Burk grafiginde cizilirse dogrular bir noktada kesisir Bu grafik olarak adlandirilir Pinpon mekanizmali enzimler arasinda bazi oksidoreduktazlar tiyoredoksin peroksidaz gibi asilnoraminat citidililtransferaz gibi ve tripsin ve gibi sayilabilir Serin proteazlar yaygin ve cesitli uyelere sahib bir enzim ailesidir aralarinda sindirici enzimler tripsin kimotripsin ve elastaz kan pihtilasma kaskadinin bazi enzimleri ve digerleri sayilabilir Serin proteazlarda E araurunu bir asil enzim turudur aktif bolge serin kalinitisinin bir protein substratinin peptit bagina saldirisi sonucu meydana gelir Kemotripsin mekanizmasini gosteren bir kisa animasyona bir baglanti surada verilmistir d Michaelis Menten disi kinetiklerSigmoid kinetik bgosteren bir enzim reaksiyonu icin doyum egrisi Bazi enzimlerin v S grafigi seklindedir bu sekilli grafik substratin aktif bolgeye baglanmasinda oldugunun belirtisidir Bu demektir ki bir substratin baglanmasi sonraki substrat molekullerinin baglanmasina etki eder Bu davranis birbiriyle etklesen birkac aktif bolgenin oldugu multimerik proteinlerde yaygindir Kooperasyon mekanizmasi hemoglobinde gorulen baglanma surecine benzer bir substratin aktif bolgeye baglanmasi diger aktif bolgelerin substrat molekullere olan afinitesini degistirir Pozitif kooperatiflik durumunda ilk substratin baglanmasi sonucu diger substratlarina olan afinite ilgi artar Negatif kooperatiflikte ise ilk substratin baglanmasi diger substratlara olan afinitenin azalmasina neden olur Allosterik enzimlere ornek olarak memeli tirozil tRNA sentetaz negatif kooperatiflik gosterir bakteryel ve pozitif kooperatiflik gosterir sayilabilir Kooperatiflik oldukca yaygindir ve substrat konsantrasyonundaki degisiklige bagli olarak enzimlerin tepkilerinin duzenlemeye yarar Pozitif kooperatiflik enzimleri S ye daha duyarli kilar ve dar bir konsantrasyon araliginda enzim aktivitesinin buyuk degisklik gostermesini saglar Buna karsin negatif kooperatiflik enzimleri S de kucuk degiskliklere az duyarli kilar Michaelis Menten kinetigine uymayan sistemlerdeki kooperatiflik derecesini nicel olarak betimlemek icin kullanilir Turetilmis Hill katsayisi n substratin bir aktif bolgeye baglanmasinin substratin diger aktif bolgelere baglanmasinin ne kadar etkilediginin olcutudur Hill kasayisi lt 1 olmasi negatik kooperatiflik Hill katsayisi gt 1 olmasi pozitif belirtir Sabit hal oncesi kinetikSabit hal oncesi ilerleme egrisi bir enzim reaksiyonundaki cogusma evresini Ing burst phase gostermektedir Bir enzim substrati ile karistirlmasindan sonraki ilk anda henuz bir urun olusmamis ve yoktur Bunu takip eden milisaniylerin arastirmasi sabit hal oncesi kinetik veya cogusma kinetigi Ingilizce Burst kinetics olarak adlandirilir Dolayisiyla sabit hal oncesi kinetigi ulasilana dek enzim substrat araurunlerinin ES veya E gibi olusumu ve tukenisi ile ilgilidir Bu yaklasim ilk defa tarafindan katalizlenen hidroliz reaksiyonuna uygulanmistir Genelde bir araurunun tespiti bir enzimin takip ettigi mekanizmayi arastirmada cok onemli bir delildir Ornegin yukarida gbelirtilen pinpon mekanizmasinda hizli kinetik olcumler bir P urununun enzimden salinmasini ve degisime ugramis bir E enzim araurununun olusumu izlenebilir Kimotripsin durumunda aktif bolgedeki nukleofilik serin substrata saldirarak bir asil enzim araurunu meydana gelir Sagdaki resimde reaksiyonun ilk birkac saniyesi sirasinda enzim hizla E olusturur Sonra sabit hale yaklastikca reaksiyon yavaslar Reaksiyonun bu hizli cogusma evresi enzimdeki tek bir devinime karsilik gelir Dolayisiyla bu patlama sirasinda salinan urun miktari grafikte y eksenindeki kesim noktasina karsilik gelir ayni zamanda reaksiyondaki fonksiyonel enzim miktarina esittir Kimyasal mekanizmaEnzim kinetiginin olcumunde onemli bir amac enzim reaksiyonunun kimyasal mekanizmasinin yani substratin urune donusturulmesindeki kimyasal adimlar dizisinin belirlenmesidir Yukarida anlatilan bu kinetik yaklasim ile araurunlerin ne hizla olustugu ve birbirine donustugunu gosterir ama bu ara urunlerin tam olarak ne oldugunu tespit edemez Farkli cozelti sartlarinda yapilan veya biraz modifiye edilmis enzim veya substratlar uzerinde yapilan kinetik olcumler cogu zaman bu kimyasal mekanizmanin anlasilmasina yardimci olur cunku hiz belirleyici adim veya reaksiyon araurunlerini ortaya cikarir Ornegin bir hidrojen atomuna olan bir kovalent bagin kirilmasi sik gorulen bir hiz belirleyici adimdir Olasil hidrojen transferlerinden hangisinin hiz belirleyici oldugu her bir hidrojeni onun kararli izotopu olan doteryum ile degistirmenin kinetik etkilerini olculerek gosterilebilir dolayi doteryum baglarinin kirilmasi hidrojene olan kovalent baglarin kirilmasindan daha zordur bu yuzden kritik hidrojen degistirildiginde reaksiyon hizi degisir Baska izotop substitusyonlari ile de 13C 12C ve 18O 16O benzer etkilerin olculmesi mumkundur ama bu etkiler ayni derecede bariz degildir Izotoplar substrat molekulunun farkli kisimlarinin kaderini ortaya cikarmak icin de kullanilabilir Ornegin bazen nihai urundeki bir oksijenin kaynagini ayirt etmek zor olabilir cunku bu atom sudan veya substrattan geliyor olabilir Hangisi oldugunu belirlemek icin reaksiyona katilan cesitli molekullere oksijenin kararli izotopu olan 18O sistematik olarak substitue edilir ve urunde bu izotopun varligina bakilir Kimyasal mekanizmanin anlasilmasi icin kullanilabilen diger yontemler kinetik ve izotop etkileri farkli pH sartlarinda enzime baglanan metal iyonlari veya diger kofaktorleri degistirerek korunmus amino asit kalintilarinin ile veya substrat analoglarinin varliginda enzimin davranisini incelemektir Enzim inhibisyonu ve aktivasyonuTersinir enzim inhibitorleri icin kinetik sema Enzim inhibitorleri enzim aktivitesini azaltan veya ortadan kaldiran mpolekullerdir buna karsin enzim aktivatorleri enzimlerin katalitik aktivitesini artiran molekullerdir Bu etkilesimler ya tersinir yani inhibitorunun giderilmesi enzim aktivitesini geri getirir veya tersinmez olabilir yani inhibitor temelli olarak enzimi inaktive eder Tersinir inhibitorler Geleneksel olarak tersinir enzim inhibitorleri Km ve Vmax uzerindeki etkilerine bagli olarak yarismali Ingilizce competitive sinirli yarismali non competitive yarismasiz uncompetitive veya karisik inhibisyon olarak siniflandirilmislardir Bu farkli etkiler inhibitorun ya E enzimine ya ES enzim substrat kompleksine ya da her ikisine birden baglanmasinin sonucudur sagdaki sekil ve asagidaki tabloda gosterildigi uzere Inhibitorun tipine ayirt etmek icin inhibitor konsantrasyonuna bagli olarak enzim kinetigi incelenir Bu dort tip inhibitor inhibitor konsantrasyonuna bagli olarak farkli ve grafikleri olusturur Sadelik icin ilgili denklemlerde su iki sembol kullanilir a 1 I Ki displaystyle alpha 1 frac mbox I K i and a 1 I Ki displaystyle alpha prime 1 frac mbox I K i prime Burada Ki ve K i sirasiyla enzime ve enzim substrat kompleksinin ayrisma sabitleridir Tersinir bir inhibitorun varliginda asagidaki tipik durumlar icin gosterildigi uzere enzimin gorunen Km ve Vmax degerleri sirasiyla a a Km ve 1 a Vmax olur Inhibisyon tipi Gorunur Km Gorunur VmaxKi sadece a 1 displaystyle alpha prime 1 yarismali Kma displaystyle K m alpha Vmax displaystyle V max Ki sadece a 1 displaystyle alpha 1 yarismasiz Kma displaystyle frac K m alpha prime Vmaxa displaystyle frac V max alpha prime Ki Ki a a displaystyle alpha alpha prime sinirli yarismali Km displaystyle K m Vmaxa displaystyle frac V max alpha prime Ki Ki a a displaystyle alpha neq alpha prime karisik Kmaa displaystyle frac K m alpha alpha prime Vmaxa displaystyle frac V max alpha prime ile enzim kinetik verilerini hiz denklemlerine uydurunca ayrisma katsayilari Ki ve K i icin yuksek dogruluklu olarak kestirmek mumkun olur Denklemlerin basit bir duzenlemesi inhibitor baglanmasi ve maksimum hiz arasindaki iliskiyi ortaya koyar Vmax1 I Ki displaystyle cfrac V max 1 cfrac I K i Vmax I KiKi displaystyle cfrac V max cfrac I K i K i Asagiya sifir ekleyince I I Vmax I Ki I Ki I displaystyle cfrac V max cfrac I K i I K i I I Ki ile bolunce Vmax11 I I Ki displaystyle cfrac V max cfrac 1 1 cfrac I I K i Vmax Vmax I I Ki displaystyle V max V max cfrac I I K i Yani reaksiyon hizinin substrat ile etkilesen enzim oranina bagli oldugu Michaelis Menten denklemine benzer sekilde substrata bagli enzim orani S S Km displaystyle cfrac S S K m inhibitore bagli enzim orani I I Ki displaystyle cfrac I I K i Inhibitorun etkisi inhibitorle etkilesen enzim populasyonunun yuzdesinin bir sonucudur Bu denklemle ilgili tek sorun inhbitor baglanmasi ile enzimin mutlak olarak inhibe oldugunu varsaymasidir oysa bu etki substrat kullaniminin 100 ila 1 in biraz fazlasi arasinda degisebilir Degisken dereceli inhibisyonu hesaba katmak icin denkleme bir delta Vmax terimi eklenir Vmax DVmax I I Ki displaystyle V max Delta V max cfrac I I K i veya Vmax1 Vmax1 Vmax2 I I Ki displaystyle V max 1 V max 1 V max 2 cfrac I I K i Bu terim sayesinde inhibitor populasyondaki enzim molekulleri ile etkilesirken kalan enzim aktivitesi tanimlananbilir Ayrica bu terimin denkleme dahil edilmesinin bir avantaji ikincil Vmax terimi ilk terimden daha buyuk olmasi halinde aktivasyon olasiliginin da hesaplanmasini mumkun kilmasidir Aktivasyon olasiligini hesaba katmak icin Inhibitor icin kullanilan I harfi yerine asagida X olarak gosterilen bir modifikator kavramina gecilebilir Vmax1 Vmax1 Vmax2 X X Kx displaystyle V max 1 V max 1 V max 2 cfrac X X K x Bu terminoloji Michaelis Menten denklemineki maksimal hiz ile ilskili kinetik etkileri daha basit bir sekilde ele almaya saglamasina karsin Km ile iliskili etkileri tarif etmekte ortaya cikabilen potansiyel problemleri de ortaya cikarir Enzimin substratina afinitesi ile ilgili olan Km cogu durumda enzim inhbitor etkilesiminin sonucu olarak enzimin baglanma yerindeki potansiyel degisikliklerle iliskili olmalidir Bu yuzden Vmax yi module eden yukaridaki benzer bir terim olmasi cogu durum icin uygun olurdu Km1 Km1 Km2 X X Kx displaystyle K m 1 K m 1 K m 2 cfrac X X K x Tersinmez inhibitorler Enzim inhibitorleri enzimleri tersinmez olarak da inaktive edebilirler genelde aktif bolgedeki amino asit kalintisini degisime ugratarak Kendilerini de yok ettikeri icin intihar substrati olarak dusunulebilecek olan bu inhibitorler reaksiyon sirasinda ussel bir azalma gosterirler ve konsantrasyona bagli olarak enzime olan etkileri genelde doyum ozelligi gosterir Doyum noktasinin altinda bu reaksiyonlar inhibitor icin birinci derece reaksiyon kinetigi gosterirler Kataliz mekanizmasiReaksiyon koordinatina gore enerji degisimi gecis halinin bir enzim tarafindan stabilize edilmesini gosterir Enzim substrat etkilesimi ile ilgili favori model induklenmis uyum modelidir Bu modele gore enzim ile substrat arasindaki ilk etkilesimin zayiftir ancak bu zayif etkilesimler enzimde konformasyonel degisiklikler dogurur indukler ve bunun sonucu olarak baglanma guclenir Bu konformasyonel degisiklikler ayrica aktif bolgedeki katalitik amino asit kalintilarini substratta reaksiyon sonucu degisecek kimyasal baglarin yakinina getirir Konformasyonel degisimler veya Ing dual polarisation interferometry ile olculebilir Baglanma olduktan sonra kataliz mekanizmalari sayesinde reaksiyon icin alternatif kimyasal yolaklar saglanarak reaksiyonun gecis halinin enerjisi alcalir Bunun olmasini saglayan kataliz mekanizmalari arasinda bag gerginligi yoluyla kataliz yakinlik ve dogrultu ile kataliz aktif bolge proton vericileri veya alicilari ile kataliz kovalent kataliz ve kuvantum tunellemesi sayilabilir Enzim kinetigi olcumleri bir enzim tarafindan hangi kataliz yolunun kullanildigini kanitlayamaz Ancak bazi kinetik veriler baska yontemlerle dogrulanabilecek olasiliklari one surebilir Ornegin sabit hal oncesi cogusmali pinpon kinetigi gosteren bir reaksiyon kovalent katalizin bu enzim reaksiyonunda onemli olabilecegini ima eder Alternatif olarak Vmax uzerinde kuvvetli bir pH etkisi olmasi ama Km uzerinde bir pH etksisi olmamasi katalizin olabilmesi icin aktif bolgedeki bir amino asit kalintisinin belli bir iyonizasyon halinde olmasi gerektigine isaret edebilir Ayrica bakinizDipnotlara b g d Kaynakca Kitap kaynagi yazar Wrighton Mark S Ebbing Darrell D baslik General chemistry yayinci Houghton Mifflin yer Boston yil 1993 isbn 0 395 63696 5 basim 4 4 Danson Michael Eisenthal Robert 2002 Enzyme assays a practical approach Oxford Oxfordshire Oxford University Press ISBN 0 19 963820 9 KB1 bakim Birden fazla ad yazar listesi link Xie XS Lu HP Haziran 1999 J Biol Chem 274 23 ss 15967 70 doi 10 1074 jbc 274 23 15967 PMID 10347141 31 Mayis 2009 tarihinde kaynagindan arsivlendi Erisim tarihi 13 Temmuz 2011 Lu H 2004 Single molecule spectroscopy studies of conformational change dynamics in enzymatic reactions Current pharmaceutical biotechnology 5 3 ss 261 9 doi 10 2174 1389201043376887 PMID 15180547 Schnell J Dyson H Wright P 2004 Structure dynamics and catalytic function of dihydrofolate reductase Annual review of biophysics and biomolecular structure Cilt 33 ss 119 40 doi 10 1146 annurev biophys 33 110502 133613 PMID 15139807 KB1 bakim Birden fazla ad yazar listesi link Gibson QH 1969 Rapid mixing Stopped flow Methods Enzymol Cilt 16 ss 187 228 doi 10 1016 S0076 6879 69 16009 7 Duggleby RG 1995 Analysis of enzyme progress curves by non linear regression Methods Enzymol Cilt 249 ss 61 90 doi 10 1016 0076 6879 95 49031 0 PMID 7791628 Murray JB Dunham CM Scott WG Ocak 2002 A pH dependent conformational change rather than the chemical step appears to be rate limiting in the hammerhead ribozyme cleavage reaction J Mol Biol 315 2 ss 121 30 doi 10 1006 jmbi 2001 5145 PMID 11779233 KB1 bakim Birden fazla ad yazar listesi link Michaelis L and Menten M L Kinetik der Invertinwirkung Biochem Z 1913 49 333 369 Ingilizce cevirisi 9 Mayis 2008 tarihinde Wayback Machine sitesinde Accessed 6 April 2007 Stroppolo ME Falconi M Caccuri AM Desideri A 2001 Superefficient enzymes Cell Mol Life Sci 58 10 ss 1451 60 doi 10 1007 PL00000788 PMID 11693526 KB1 bakim Birden fazla ad yazar listesi link Walsh R Martin E Darvesh S A method to describe enzyme catalyzed reactions by combining steady state and time course enzyme kinetic parameters Biochim Biophys Acta 2010 Jan 1800 1 5 Jones ME 1 Ocak 1992 Analysis of algebraic weighted least squares estimators for enzyme parameters Biochem J 288 Pt 2 ss 533 8 PMC 1132043 2 PMID 1463456 a b Tseng SJ Hsu JP Agustos 1990 A comparison of the parameter estimating procedures for the Michaelis Menten model J Theor Biol 145 4 ss 457 64 doi 10 1016 S0022 5193 05 80481 3 PMID 2246896 Bravo IG Busto F De Arriaga D ve digerleri Eylul 2001 A normalized plot as a novel and time saving tool in complex enzyme kinetic analysis Biochem J 358 Pt 3 ss 573 83 PMC 1222113 2 PMID 11577687 KB1 bakim Digerlerinin yanlis kullanimi link KB1 bakim Birden fazla ad yazar listesi link Almaas E Kovacs B Vicsek T Oltvai ZN Barabasi AL Subat 2004 Global organization of metabolic fluxes in the bacterium Escherichia coli Nature 427 6977 ss 839 43 doi 10 1038 nature02289 PMID 14985762 KB1 bakim Birden fazla ad yazar listesi link Reed JL Vo TD Schilling CH Palsson BO 2003 An expanded genome scale model of Escherichia coli K 12 iJR904 GSM GPR Genome Biology 4 9 ss R54 doi 10 1186 gb 2003 4 9 r54 PMC 193654 2 PMID 12952533 KB1 bakim Birden fazla ad yazar listesi link Denklemin turetilmesi icin bakiniz here Dirr H Reinemer P Huber R Mart 1994 X ray crystal structures of cytosolic glutathione S transferases Implications for protein architecture substrate recognition and catalytic function Eur J Biochem 220 3 ss 645 61 doi 10 1111 j 1432 1033 1994 tb18666 x PMID 8143720 KB1 bakim Birden fazla ad yazar listesi link Stone SR Morrison JF Temmuz 1988 Dihydrofolate reductase from Escherichia coli the kinetic mechanism with NADPH and reduced acetylpyridine adenine dinucleotide phosphate as substrates Biochemistry 27 15 ss 5493 9 doi 10 1021 bi00415a016 PMID 3052577 Fisher PA 1994 Enzymologic mechanism of replicative DNA polymerases in higher eukaryotes Prog Nucleic Acid Res Mol Biol Cilt 47 ss 371 97 doi 10 1016 S0079 6603 08 60257 3 PMID 8016325 Akerman SE Muller S Agustos 2003 2 Cys peroxiredoxin PfTrx Px1 is involved in the antioxidant defence of Plasmodium falciparum Mol Biochem Parasitol 130 2 ss 75 81 doi 10 1016 S0166 6851 03 00161 0 PMID 12946843 20 Subat 2008 tarihinde kaynagindan Erisim tarihi 13 Temmuz 2011 Bravo IG Barrallo S Ferrero MA Rodriguez Aparicio LB Martinez Blanco H Reglero A Eylul 2001 Kinetic properties of the acylneuraminate cytidylyltransferase from Pasteurella haemolytica A2 Biochem J 358 Pt 3 ss 585 98 PMC 1222114 2 PMID 11577688 KB1 bakim Birden fazla ad yazar listesi link Kraut J 1977 Serine proteases structure and mechanism of catalysis Annu Rev Biochem Cilt 46 ss 331 58 doi 10 1146 annurev bi 46 070177 001555 PMID 332063 olu kirik baglanti Ricard J Cornish Bowden A Temmuz 1987 Co operative and allosteric enzymes 20 years on Eur J Biochem 166 2 ss 255 72 doi 10 1111 j 1432 1033 1987 tb13510 x PMID 3301336 Ward WH Fersht AR Temmuz 1988 Tyrosyl tRNA synthetase acts as an asymmetric dimer in charging tRNA A rationale for half of the sites activity Biochemistry 27 15 ss 5525 30 doi 10 1021 bi00415a021 PMID 3179266 Helmstaedt K Krappmann S Braus GH Eylul 2001 Allosteric regulation of catalytic activity Escherichia coli aspartate transcarbamoylase versus yeast chorismate mutase Microbiol Mol Biol Rev 65 3 ss 404 21 table of contents doi 10 1128 MMBR 65 3 404 421 2001 PMC 99034 2 PMID 11528003 11 Haziran 2011 tarihinde kaynagindan Erisim tarihi 13 Temmuz 2011 KB1 bakim Birden fazla ad yazar listesi link Schirmer T Evans PR Ocak 1990 Structural basis of the allosteric behaviour of phosphofructokinase Nature 343 6254 ss 140 5 doi 10 1038 343140a0 PMID 2136935 Hill A V The possible effects of the aggregation of the molecules of haemoglobin on its dissociation curves J Physiol Lond 1910 40 iv vii Hartley BS Kilby BA Subat 1954 The reaction of p nitrophenyl esters with chymotrypsin and insulin Biochem J 56 2 ss 288 97 PMC 1269615 2 PMID 13140189 a b Fersht Alan 1999 Structure and mechanism in protein science a guide to enzyme catalysis and protein folding San Francisco W H Freeman ISBN 0 7167 3268 8 Bender ML Begue Canton ML Blakeley RL ve digerleri Aralik 1966 The determination of the concentration of hydrolytic enzyme solutions alpha chymotrypsin trypsin papain elastase subtilisin and acetylcholinesterase J Am Chem Soc 88 24 ss 5890 913 doi 10 1021 ja00976a034 PMID 5980876 KB1 bakim Digerlerinin yanlis kullanimi link KB1 bakim Birden fazla ad yazar listesi link Cleland WW Ocak 2005 The use of isotope effects to determine enzyme mechanisms Arch Biochem Biophys 433 1 ss 2 12 doi 10 1016 j abb 2004 08 027 PMID 15581561 20 Subat 2008 tarihinde kaynagindan Erisim tarihi 5 Eylul 2008 Northrop D 1981 The expression of isotope effects on enzyme catalyzed reactions Annu Rev Biochem Cilt 50 ss 103 31 doi 10 1146 annurev bi 50 070181 000535 PMID 7023356 Baillie T Rettenmeier A 1986 Drug biotransformation mechanistic studies with stable isotopes Journal of clinical pharmacology 26 6 ss 448 51 PMID 3734135 Cleland WW 1982 Use of isotope effects to elucidate enzyme mechanisms CRC Crit Rev Biochem 13 4 ss 385 428 doi 10 3109 10409238209108715 PMID 6759038 Christianson DW Cox JD 1999 Catalysis by metal activated hydroxide in zinc and manganese metalloenzymes Annu Rev Biochem Cilt 68 ss 33 57 doi 10 1146 annurev biochem 68 1 33 PMID 10872443 Kraut D Carroll K Herschlag D 2003 Challenges in enzyme mechanism and energetics Annu Rev Biochem Cilt 72 ss 517 71 doi 10 1146 annurev biochem 72 121801 161617 PMID 12704087 KB1 bakim Birden fazla ad yazar listesi link Leatherbarrow RJ Aralik 1990 Using linear and non linear regression to fit biochemical data Trends Biochem Sci 15 12 ss 455 8 doi 10 1016 0968 0004 90 90295 M PMID 2077683 Walsh R Martin E Darvesh S A versatile equation to describe reversible enzyme inhibition and activation kinetics modeling beta galactosidase and butyrylcholinesterase Biochim Biophys Acta 2007 1770 733 46 Koshland DE Subat 1958 Application of a Theory of Enzyme Specificity to Protein Synthesis Proc Natl Acad Sci U S A 44 2 ss 98 104 doi 10 1073 pnas 44 2 98 PMC 335371 2 PMID 16590179 Hammes G 2002 Multiple conformational changes in enzyme catalysis Biochemistry 41 26 ss 8221 8 doi 10 1021 bi0260839 PMID 12081470 Sutcliffe M Scrutton N 2002 A new conceptual framework for enzyme catalysis Hydrogen tunnelling coupled to enzyme dynamics in flavoprotein and quinoprotein enzymes Eur J Biochem 269 13 ss 3096 102 doi 10 1046 j 1432 1033 2002 03020 x PMID 12084049 8 Kasim 2006 tarihinde kaynagindan Erisim tarihi 13 Temmuz 2011 Konuyla ilgili yayinlarGiris duzeyi Kitap kaynagi yazar Cornish Bowden Athel baslik Fundamentals of enzyme kinetics yayinci Portland Press yer Londra yil 2004 isbn 1 85578 158 1 basim 3 3 Ingilizce Stevens Lewis Price Nicholas C 1999 Fundamentals of enzymology the cell and molecular biology of catalytic proteins Oxford Oxfordshire Oxford University Press ISBN 0 19 850229 X KB1 bakim Birden fazla ad yazar listesi link Ingilizce Bugg Tim 2004 Introduction to Enzyme and Coenzyme Chemistry Cambridge MA Blackwell Publishers ISBN 1 4051 1452 5 Ingilizce Ileri duzey Segel Irwin H 1993 Enzyme kinetics behavior and analysis of rapid equilibrium and steady state enzyme systems New bas New York Wiley ISBN 0 471 30309 7 Ingilizce Fersht Alan 1999 Structure and mechanism in protein science a guide to enzyme catalysis and protein folding San Francisco W H Freeman ISBN 0 7167 3268 8 Ingilizce Santiago Schnell Philip K Maini 2004 PDF Comments on Theoretical Biology Cilt 8 ss 169 87 doi 10 1080 08948550302453 21 Subat 2006 tarihinde kaynagindan PDF arsivlendi Erisim tarihi 13 Temmuz 2011 Ingilizce Walsh Christopher 1979 Enzymatic reaction mechanisms San Francisco W H Freeman ISBN 0 7167 0070 0 Ingilizce Cleland William Wallace Cook Paul 2007 Enzyme kinetics and mechanism New York Garland Science ISBN 0 8153 4140 7 KB1 bakim Birden fazla ad yazar listesi link Ingilizce Dis baglantilarBir enzim olcumunun animasyonu 6 Ekim 2011 tarihinde Wayback Machine sitesinde Shows effects of manipulating assay conditions MACiE 8 Agustos 2011 tarihinde Wayback Machine sitesinde Enzim mekanizmalari veritabani ENZYME6 Ekim 2006 tarihinde Wayback Machine sitesinde Expasy enzim adlandirma sistemi veritabani Enzim katalitik mekanizmalari veritabani BRENDA4 Kasim 2011 tarihinde Wayback Machine sitesinde Kapsamli bir enzim veritabani substratlar inhibitorler ve reaksiyon cizimleri bulunmaktadir Reaksiyon kinetkileri veritabani Birkac enzim reaksiyonun mekanizmasinin animasyonu Enzim kinetigindeki kavramlar ve terminolojinin kapsamli bir aciklamasi Enzim kinetigi hakkinda cevrimici kolay anlasilir bir egitsel Enzim kinetigi animasyonlu egitsel 24 Temmuz 2011 tarihinde Wayback Machine sitesinde
 Azərbaycanca
Azərbaycanca Беларускі
Беларускі Dansk
Dansk Deutsch
Deutsch Española
Española Français
Français Indonesia
Indonesia Italiana
Italiana 日本語
日本語 Қазақ
Қазақ Lietuvos
Lietuvos Nederlands
Nederlands Português
Português Русский
Русский සිංහල
සිංහල แบบไทย
แบบไทย Türkçe
Türkçe Українська
Українська 中國人
中國人 United State
United State Afrikaans
Afrikaans